의연방05) 코호트연구와 단면조사연구
Cohort study & Cross-sectional study
📜 제목으로 보기✏마지막 댓글로
- 학습개요
- Cohort study
- 연구디자인과 cohort study
- 코호트의 목적: 위험요소 - 질병발생 상관관계 탐구
- 예제로 보는 cohort study 속 위험요소-질병발생 상관관계 by RR, OR
- R 실습(상관관계를 위한 RR,OR,신뢰구간,독립성검정까지 반영한 상관관계)
- confounding까지 고려한 코호트 속 위험요소-질병간 상관관계
- R 실습(confounding or interaction까지 고려한 상관관계)
- 연구에서 Confounding과 Interaction(effect modifier)를 만났을 때 설계
- 2분형 결과변수의 코호트 분석 제한점
- 코호트 스터디 논문예제
- Cross-sectional Study
- 정리하기
- R 고급 실습(cohort study with dplyr-pipeline, broom-tidy) 포함됨
-
Cohort study의 정의를 설명할 수 있다.
-
Cohort study에서 RR과 OR을 추정하고, 위험요소와 질병의 독립성을 검정할 수 있다.
-
로지스틱 회귀분석을 이용하여 confounding과 interaction에 대한 분석을 수행할 수 있다.
-
Cross-sectional study의 정의를 설명하고, cohort study와의 차이점을 설명할 수 있다.
-
Cohort study : 연구자가 위험요소에 노출된 그룹과 노출되지 않은 그룹을 일정기간 동안 추적관찰하여, 두 그룹 간 질병 발생률(incidence)이나 질병에 의한 사망률 등을 비교하는 연구
-
Mantel-Haenszel 방법 : Confounder값 별로 나눈 stratum에서 각각 구한 OR(또는 RR)을, 그 OR의 분산의 역수를 weight로 준 weighted average(가중평균)으로 종합하는 방법
-
Logistic regression : 결과변수(반응변수, 종속변수)가 이분형인 회귀분석
-
Cross-sectional study : 위험요소 노출여부와 질병유무를 한 시점에 동시에 조사하는 연구
- 연구종류에는
-
대상자에게 뭔가를 가하는
Experimental(익스페리멘탈) study과 -
대상자에게 뭔가를 하지않고 관찰만하는
Observational(옵져베이셔널) study가 있다.
-
대상자에게 뭔가를 가하는
- 대상자에게 뭔가를 가하지 않는
옵져베이셔널 스터디(관찰연구)에는-
전향적인 연구인 Prospective study와 후향적인 연구인 Retrospective study가 있다.
- 전향적인 연구: 연구시점부터 무슨일이 일어나는지 관찰한다.
- 후향적인 연구: 다 끝난 뒤에서 무슨일이 일어났었는지 관찰한다.
전향적인 관찰연구(→) 중 적어도 2시점 이상을 관찰하는 연구를Cohort Study(=Longitudinal Study)라 한다.전향이든, 후향이든 관찰연구(→, ←) 중 딱 1시점에서만 관찰하는 연구를Cross-sectional Study(=? Case-control study?)라 한다.
-
전향적인 연구인 Prospective study와 후향적인 연구인 Retrospective study가 있다.
-
연구자가
위험요소 1개에 대해노출O(group)/ 노출X(group)의 그룹을 나누고- 위험요소 뿐만 아니라
치료법 O/Xor환자 특정 특성 O/X(ex나이)의 2그룹으로 나눌 수 있다.- ex> 남성/여성
- 실제로는 2그룹 뿐만 아니라 3그룹 이상도 나눌 수 있다.
- ex> 20-40세 / 40-60세 / 60세이상의 3그룹
- 위험요소 뿐만 아니라
-
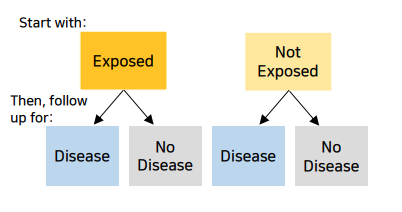
일정기간 동안 추적 관찰하여,그룹간 질병 발생률(incidence)나 질병 사망률(또는 다른 이벤트) 등을 비교하는 연구
- 연구
시작 시점에 위험요소(치료법, 특성)노출O 그룹 vs 노출X 그룹을 먼저 나누고->각 그룹별 질병 발생/사망률 추적 관찰- 예> 방사능 노출이라는 위험요소에 대해, 노출시 아웃컴이 어떻게 변하는가?
-
원전사고가 있는 지역 = exposed group과원전사고가 없지만, 비슷한 지역 = not exposed group그룹을 먼저 나누고 - 질병 발생률이나 사망률을 follow up
-
- 예> 방사능 노출이라는 위험요소에 대해, 노출시 아웃컴이 어떻게 변하는가?
- 연구 시작 시점에
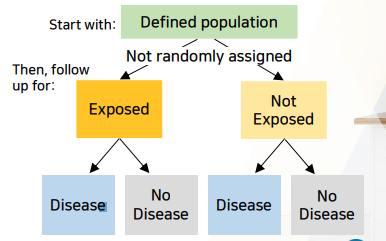
아직 위험요소(치료법, 특성)에 노출X 집단만 선택해놓고 ->위험요소 노출여부O/X관찰 ->질병발생률다시 관찰(2가지를 관찰)- 이 때, 노출여부O/X는 랜덤화 하지않고, 자연스럽게 그룹이 나뉘게 한다.
- 연구 시작시점(방법1) or 연구기간(방법2) 중
위험요소 노출여부가 먼저관측 -> 그 이후에새로 발생한 질병관측하기 때문에-
위험요소 -> 질병발생의 선후가 정해져 있어서 질병발생 시점이 명확 -> incidence(질병발생 속도와 관련) 추정이 가능 -
위험요소 -> 질병발생의 선후가 정해져 있어서 인과관계 추론에 결정적 단서 -
위험요소 -> 질병발생의 선후가 정해져 있어서 위험요소 노출O/X이후 나올 수 있는 여러 질병들을 동시에 연구 가능하다.(타겟질병말고도 다른 질병이 나타나면 연구가능)
-

- 질병 종류에 따라서 너무 오래걸리거나 비용이 많이 들 수 있다.
-
위험요소 -> 질병발생의 선후가 정해져 있어서 질병 발생할때까지 계속 기다려야한다. -
위험요소 -> 질병발생의 선후가 정해져 있어서 희귀병은 기다려도 몇 명 발생안하므로, 많은 수의 연구대상자가 확보되어야하고, 계속 follow up해야한다-
대안:
retrospective cohort study- 전통적인 코호트 스터디는 전향적 연구( 연구시작 -> 노출O/X그룹 -> 질병발생률 관찰)인데
- 요즘은 레트로스펙티브 코호트 스터디도 소개되고 있다.
-
연구시작 시점 이후발병하는 질병을 follow 하는 것은 동일하나, -
연구시작 시점 이전위험요소 노출O/X를 <기록(데이터),기억(인터뷰)>를 통해 결정하는 것- 질병발생률은 prospective / 위험요소 노출여부는 retrospective로서 2가지가 섞여있는 디자인이라고도 볼 수 있다.
- 위험요소 노출O/X를 기다렸다가 나누었다가 할 필요가 없이 바로 질병발생률만 관찰하므로 전체적인 연구기간이 짧아진다.
-
대안:
-
위험요소 -> 질병발생의 선후가 정해져 있어서 질병 발생할때까지 계속 기다리다가 위험요소 노출여부가 중간에 바뀌어버릴 수 있다.- 예> 흡연으로 위험요소 노출O였다가, 연구 중간에 금연해서 not/un exposed group으로 가버릴 수 있다
- 대안: 위험요소 노출여부를 여러번 측정 -> longitudinal study(여러번 측정시)
-

- 전형적이지 않게, 1948년 연구 시작으로 지금까지 연구중
- 위험요소가 무엇인지 모를 때 -> 심혈관계 질환 발생에 어떤 것들이??
- 위험요소라는 결정적인 증거가 없었을 때, 위험요소들을 모아보기 위해 연구 시작
- 식생활, 비만 등
- 맨 첨 시작할 때는 위험요소노출O/X 이후 -> 질병발생까지 기다리는 시간이 오래 걸리니 30세 이상부터 연구 -> 5000명 정도 시작
- 자식세대인 offspring cohort는 5000명 정도 follow 중
- 3세대는 80세 미만으로 400명 정도가 follow 중

-
이 상관관계를 탐구하기 위해 사용하는 메져가 무엇이 있었는가?
- RR 랠러티브 리스크 for 상관관계
- OR 오즈 레이시오 for 상관관계
- attributable risk for 상관관계
-
RR과 OR을 코호트 스터디에선 어떻게 측정하는지 알아보자

- RR을 구하려면
-
exposed risk(위험요소 노출 O group에서의 질병발생 확률)을p1로 구한다. -
unexposed risk(위험요소 노출 X group에서의 질병발생 확률)을p0로 구한다. -
RR는 p1/p0로위험요소 노출X 그룹의 risk분에노출O 그룹의 risk로 위험요소노출 그룹별로 구한 risk들의 비율이다.
-
- OR을 구하려면
-
exposed riskp1을 구한다 -
unexposed riskp0을 구한다 -
exposed Odds를 p1/1-p1으로 구한다. -
unexposed Odds를 p0/1-p0으로 구한다. -
OR는위험요소 노출X 그룹의 odds분에노출O 그룹의 odds로 위험요소노출 그룹별로 구한 odds의 비율이다.
-
-
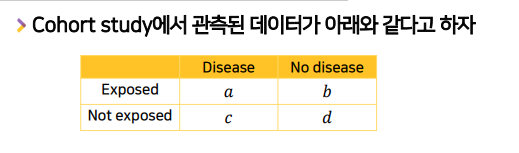
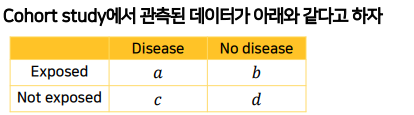
표에서 row/col을 보고 질병발생여부보다
위험요소 노출 여부가 있는 row별로 확인한다.- 위험요소 노출O -> 질병발생 a명 / 질병발생안한 사람 b명
- 위험요소 노출X -> 질병발생 c명 / 질병발생안한 사람 d명
-
RR은 각
위험요소 노출O/X group별 risk의 비율이므로 각 group별 risk를 구한다.- exposed risk(p1) : a/a+b
- unexposed risk(p0) : c/c+d
- rr : p1/p0 = a/a+b / c/c+d

-
표에서 row/col을 보고 질병발생여부보다
위험요소 노출 여부가 있는 row별로 확인한다. 2.OR는 각위험요소 노출O/X group별 odds의 비율이므로 각 group별 odds를 구해야한다.- 각 위험요소 노출여부 group별 odds는
그룹별 risk(p)부터 구한뒤 ->그룹별 odds(p/1-p)를 구해야한다.- exposed odds
- exposed risk(p1) : a/a+b
- exposed odds(p1/1-p1) : a/a+b / (1 - a/a+b) = a/b
- unexposed odds
- unexposed risk(p0) : c/c+d
- unexposed odds(p0/1-p0) : c/c+d / (1 - c/c+d) = c/d
- exposed odds
- OR = exposed odds / unexposed odds = a/b / c/d = ad/bc

- 각 위험요소 노출여부 group별 odds는
-
구하고 보니
위험요소여부-질병발생여부 표에서 cross-product하면,odds ratio를 바로 구할 수 도 있다.- 이 때, a는 위험요소노출o->질병o, d는 위험요소노출x->질병x 이다.

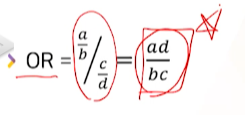
- 이 때, a는 위험요소노출o->질병o, d는 위험요소노출x->질병x 이다.
- 표를 보고 위험요소 노출O/X(row for group) -> 질병 발생여부(col) 순서로 확인한다.
- 아~ 위험요소 2개의 interaction문제는 아니구나
- 아~
위험요소 1개 O/X별 -> 질병발생 O/X 의 관계이므로 -> 상관관계가 목적이고 -> RR, OR로 계산하겠구나. 그럴려면위험요소 O/X를 group으로 나눠서 risk/odds를 계산하겠구나
-
RR를
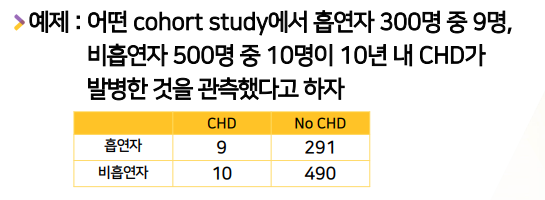
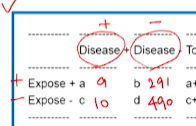
각 위험요소 노출여부 그룹별(→row별) risk 비율로 구한다.- RR = exposed risk(p1) / unexposed risk(p0) = (9/9+291) / (10/10+490) = 9/300 / 10/500 = 1.5
-
OR는 위험요소노출여부-질병여부 표에서
ad/bc의 cross-product로 빠르게 구한다- 원래는
각 위험요소 노출여부 그룹별(→row별) odds(risk/1-risk) 비율로 구해야한다. - OR = a(oo)d(xx) / bc = 9 x 490 / 291 x 10 = 1.52

- 원래는
- RR, OR을 계산하는 이유는 위험요소 - 질병의
상관관계의 크기를 보기 위함이고, - RR, OR의 점추정에 대한 불확실성 때문에
상관관계의 크기 신뢰구간까지 더해준다. -
위험요소 - 질병의
상관관계가 통계적으로 유의한지를2개가 독립이 아닌지로 통계적 검정까지 해줘야한다- 위험요소 - 질병 상관관계는 RR, OR로 구한다며... 근데..
신뢰구간과독립아닌지 검정까지해줘야해?
- 위험요소 - 질병 상관관계는 RR, OR로 구한다며... 근데..
-
상관관계의 크기(RR, OR) 및 신뢰구간을 구했다면, 그래서 이것이 유의한 상관관계이냐를
독립이 아니냐의 통계적 검정(하이퍼 시시스 테스트)을 통해 증명한다.- 일반:
위험요소 - 질병간 상관관계가 있느냐? - 통계적 표현:
위험요소노출여부라는 변수와질병발생여부라는 변수가서로 독립이냐?(독립아니다의 대립가설을 채택해야함)
- 일반:
-
위험요소 노출여부는 2그룹 이외에 3~4그룹으로도 나눌수 있지만,
질병발생여부는 무조건 2분형이어야한다. -
위험요소여부, 질병여부 ->
2개의 범주형 변수에 대한독립성 검정은카이스퀘어 검정을 이용한다- H0(귀무가설): 독립이다. RR=1 or OR=1
- H1(대립가설): 독립이 아니다(상관관계 있다) RR != 1 or OR != 1
-
카시스퀘어 검정은 샘플작을 때 작동을 잘 안하므로
Fisher's exact Test를 이용한다
- 서로 독립이다를 수식으로 나타내면 Pij = Pi x Pj로 나타낸다.
- 첫째 범주 i, 2번째 범주 j

- 첫째 범주 i, 2번째 범주 j
- 테스트하는 검정통계량
- 귀무가설 하에서의 기대도수를 이용하여 계산된 검정통계량을 사용한다(공식)
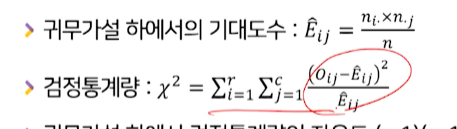
- 귀무가설 하에서의 기대도수를 이용하여 계산된 검정통계량을 사용한다(공식)
-
여러 방법이 있지만,
epiR패키지로 계산한다.- 업데이트에 민감하니, epiR패키지를 업데이를 하고 R을 최신버전으로 업그레이드까지.. 안되면 epiR 제거후 다시 설치
-
2by2 table을 입력할 때, 이미 형식이 정해져있다.
- 위험요소가 row / 질병여부가 col에 있다고 가정하고
- 위험o : 질병o(a) 질병x(b)
- 위험x : 질병o(c) 질병x(d)
- 의 순서대로
c(a, b, c, d)순서대로 입력해줘야한다.- my) 앞이 위험요소/뒤가 질병여부라 치면
row1: oo(a) ox(b) //row2: xo(c) xx(d)순으로 입력
- my) 앞이 위험요소/뒤가 질병여부라 치면
- 위험요소가 row / 질병여부가 col에 있다고 가정하고
# 아나콘다에서 epiR 검색후 r-epir 설치
library(epiR)
# 9, 291,
# 10, 490
# )
# tab <- c(9, 291, 10, 490)
# 바로 벡터만 넣어주면 안되더라... matrix nrow=2 byrow 써서 matrix를 만들어줘야함
# ?epi.2by2
tab <- matrix(c(9, 291, 10, 490), nrow = 2, byrow = TRUE)
# rownames(dat) <- c("Expose+", "Expose-");
# colnames(dat) <- c("Disease+", "Disease-");
# epi.2by2(dat = as.table(dat), method = "cross.sectional",
# conf.level = 0.95, units = 100, outcome = "as.columns")
tab
epi.2by2(dat=tab, method="cohort.count") # method 코호트.카운트가 default긴 하다.
# case-control인 경우, method옵션을 바꾸면 된다.
# -> RR ( 95% 신뢰구간이 자동 제공된다.)
# Odds ratio 1.52 (0.61, 3.77)
# -> OR ( 95% 신뢰구간)
# 신뢰구간은 wald방법으로 뽑아진 것
# 아래 attributable risk로 계산 한 것
# Attrib risk * 1.00 (-1.29, 3.29)
# Attrib risk in population * 0.38 (-1.24, 1.99)
# Attrib fraction in exposed (%) 33.33 (-62.19, 72.60)
# Attrib fraction in population (%) 15.79 (-28.34, 44.75)
# Test that odds ratio = 1: chi2(1) = 0.809 Pr>chi2 = 0.369
# 0.05보다 높아서 -> H0 독립성 보장 -> **상관관계 유의하지 않다**
- 데이터분석 단계에서 confouning해결책 3가지 중에 2가지만 제시(후속강의에서 3번째 방법 진행)
- Stratification : 각 confounder 값 별로 strata를 나누어,
각 stratum별로 상관관계를 따로 추정한다-
각 confouder값 별로 따로 추정한 상관관계를 종합하는 방법이
Mantel-Haenszel 방법- 각 stratum별 RR이나 OR을 종합
-
각 confouder값 별로 따로 추정한 상관관계를 종합하는 방법이
-
Adjustment : 기본적으로 Multivariable logistic regression를 하는 것으로 confounder를 공변량으로 넣은 회귀분석을 통해, confounder 값이 고정되어 있을 때의 X와 Y의 상관관계를 추정한다
-
Multivariable logistic regression: adjusted OR

-
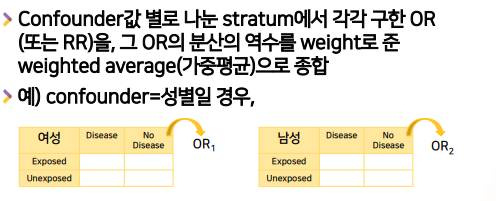
-
confounder 값별로 따로2by2 table을 만들고 -> OR(RR)을 계산한다 - 각 confounder값별로 나온 OR1,OR2 .. 를 평균을 내는데, 자신이 있는 지에 따라
분산의 역수를 weight로 준 가중 평균을 구한다.- 좀 더 계산에 자신 있는 OR에 비중을 많이 둬서 평균을 구하는데
-
(추정값에 대한) 자신 있다 없다는분산(불확실성)으로 표현된다. 분산이 크면 -> 불확실성 크다 -> 자신 없다 -> 분산의 역수 = 자신 있다 -> 분산의 역수를 weight로 줘서 자신있는 곳에 비중을 둔다.
- 예
- confoudner가 성별이라고 치면, 여성에 대한 2by2 table , 남성에 대한 2by2 table을 따로 만들고, OR도 따로 구한다.
-
OR의 평균을 분산의 역수로 가중평균을 구한다. ->
추정값에 대한 변동이 적은=추정값에 대해 불확실성이 적은=추정값에 대해 내가 더 자신있는=대체로 샘플사이즈가 더 큰 confounder의OR을 더 많이 반영할 수 있다.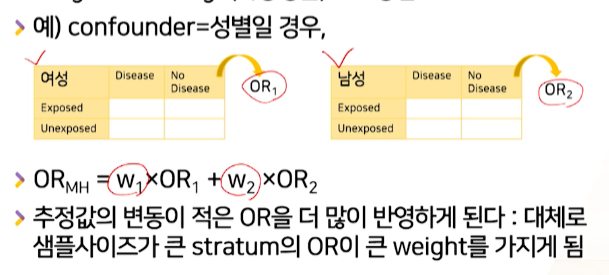
- 계산과정을 생략하고, 전체적으로 보면
- confounder 값의 갯수만큼(I개)로 2by2 table -> OR이 나오며, 계산방법을 따라서 OR를 종합할 수 있다.

- confounder 값의 갯수만큼(I개)로 2by2 table -> OR이 나오며, 계산방법을 따라서 OR를 종합할 수 있다.
# install.packages("Epi")
library(epiR)
library(Epi) # 데이터용
library(dplyr) # 데이터정리용
?nickel # lung cancer (ICDs 162 and 163), nasal cancer (ICD 160)
data(nickel) # 예제데이터
dat1 <- nickel
head(dat1)
# exposure: 위험요소 노출된 정도 -> 위험요소 그룹은 2개를 넘어선다
# age1st : 위험요소에 노출된 첫번째 나이
# icd : 어떤이유로 사망했는지 알 수 있는데, 죽지 않았으면 0
# - lung cancer는 162, 163으로 코딩되어있고 / nasal cancer 는 160으로 코딩
# ifelse()로 삼항연산자를 이용한 매핑을 한다.
dat1 <- nickel %>%
mutate(lung.cancer = ifelse(icd==162 | icd == 163, 1, 0))
head(dat1)
dat1 <- nickel %>% mutate(
lung.cancer = ifelse(icd==162 | icd == 163, 1, 0),
nasal.cancer = ifelse(icd==160, 1, 0),
)
head(dat1)
dat1 <- nickel %>% mutate(
lung.cancer = ifelse(icd==162 | icd == 163, 1, 0),
nasal.cancer = ifelse(icd==160, 1, 0),
# 연속형 변수로 표현되어있던 위험요소 노출 정도를 -> 특정값 기준으로 위험요소 노출여부(1or0)로 매핑
exposure.grp = ifelse(exposure >= 10, 1, 0),
# 위험요소노출 나이 또한 2분형 범주칼럼으로 매핑
age1st.grp = ifelse(age1st >= 25, 1, 0))
head(dat1)
dat1 <- nickel %>% mutate(
lung.cancer = ifelse(icd==162 | icd == 163, 1, 0),
nasal.cancer = ifelse(icd==160, 1, 0),
exposure.grp = ifelse(exposure >= 10, 1, 0),
age1st.grp = ifelse(age1st >= 25, 1, 0)) %>%
mutate_at(vars(lung.cancer, nasal.cancer,
exposure.grp, age1st.grp),
list(~as.factor(.)))
head(dat1)
# 질병 여부 -> nasal.cancer
# 정해진 범주형 칼럼을 table()로 만든다. -> 알아서 범주종류별로 n by m을 만들어주나보다.
# -> 만약, 값만 있다면? as.matrix( a,b,c,d , row=2, byrow=true)
# 먼저 적어준 범주형칼럼이 row로 오게 된다?
table(dat1$exposure.grp, dat1$nasal.cancer)
tab2 <- table(dat1$exposure.grp, dat1$nasal.cancer,
dnn = c("exposure.grp", "nasal.cancer"))
tab2
# 출력해보니, 위험요소/질병여부 oo -> ox 순이 아니라 xx -> xo 순으로 나온다.
# -> 코호트나 분석을 위해선, oo, ox xo xx 순으로 나타나도록
# --> 데이터 dat1 속 factor의 level순서를 재설정해줘야한다.
dat2 <- dat1 %>% mutate(lung.cancer=relevel(lung.cancer, ref="1"),
nasal.cancer=relevel(nasal.cancer, ref="1"),
exposure.grp=relevel(exposure.grp, ref="1"),
)
# age1st.grp=relevel(age1st.grp, ref="1"))
# 이놈은 나중에 confounder로 사용될 변수라서.. 딱히 1의 그룹을 첫번째그룹으로 안주어도 된다.
head(dat2)
tab2 <- table(dat2$exposure.grp, dat2$nasal.cancer,
dnn = c("exposure.grp", "nasal.cancer"))
tab2
epi.2by2(tab2)
- age1st.grp도 0or1로 매핑했었다 -> 이걸 confounder라고 가정하고, confounder값별로 나눠서 2by2 테이블을 그리는 것을
Stratification라 한다
tab3 <- table(dat2$exposure.grp, dat2$nasal.cancer, dat2$age1st.grp,
dnn = c("exposure.grp", "nasal.cancer", "age1st.grp"))
tab3
# table()에 3번째 인자를 주면, 그 값별로 1,2번째 칼럼으로 만든 table을 나눠서 보여준다.
# 3번째칼럼의 범주종류별로 3차원을 table이 완성된다고 생각하자.
epi.2by2( tab3 )
# 데이터 자체는 위에서 만든 table과 유사하나, 아래 요약글을 보면
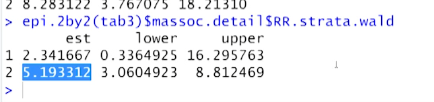
# RR, OR이 여러줄로 나타난다.( cf) rsk ratio = relative risk
# Inc risk ratio (crude) -> crude: confounder(age1st.grp)을 무시한 rr + 신뢰구간
# Inc risk ratio (M-H) -> [맨틀-핸젤 방법으로 confounder별 (다른 table->) 다른 RR을 가중평균한] RR + 신뢰구간
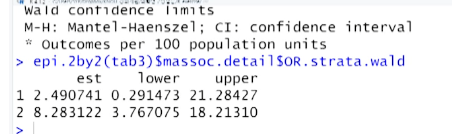
# Odds ratio (crude) 7.77 (3.85, 15.69)
# Odds ratio (M-H) 7.08 (3.45, 14.54)
# -> confounder 무시한 OR + cofounder별 OR구한 뒤 맨틀-핸젤방법으로 가중평균한 OR
# [m]easure of [assoc] iation의 detail이라는 뜻.
epi.2by2( tab3 )$massoc.detail$OR.strata.wald
epi.2by2( tab3 )$massoc.detail$RR.strata.wald
-
나는 안되네..
- confounder를 어저스트먼트(adjustment)로 보정하기 위해선
Multivaraible logistic regression이 필요하므로 logisitic regression에 대해 먼저 알아보자
- 결과 변수가 이분형(binary)인 회귀분석
- 우리는 지금 코호트 속 설명변수
위험요소 노출여부에 대한질병여부로서 결과변수를 이분형을 사용중이니 이분형 결과변수를 사용하는 logisitic regression을 적절한 회귀분석으로 사용할 수 있다- 위험요소 노출여부를 x(설명변수) , 질병
여부를y결과변수(종속변수)로 로지스틱 회귀분석 모델을 잡을 수 있는데 -
회귀식의 좌항은 ln(
질병발생O 그룹의 odds) = 1차 회귀식(독립변수의 linear combination)으로 성립한다.- 즉, 로지스틱 회귀 모델은
질병발생O의 odds에 자연로그 취한 값을 =독립변수들의 linear combination으로 설명하겠다는 뜻이 된다.
- 즉, 로지스틱 회귀 모델은
- 위험요소 노출여부를 x(설명변수) , 질병
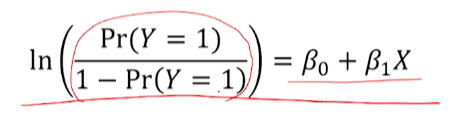

-
ln(질병O의 odds) = b0 + b1*X의 로지스틱 회귀 식에서- X=1을 대입 후, 양변에 exponentiate(e, 지수함수)을 취해주자.
- X=1이면, 위험요소 노출O의 상황이므로 risk를
p1이라고 표현한다(앞에서는 위험여부O일 때의 risk를 p1로 취급함)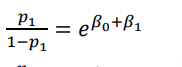
- X=1이면, 위험요소 노출O의 상황이므로 risk를
- X=0을 대입후, 위험요소 노출O의 상황이므로 risk를
p0이라 표현하고, 양변에 지수함수를 취한다.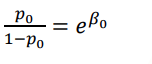
- OR(odds ratio)는 X=0일 때의 odds 분에 X=1일때의 odds (odds의 비율)이므로, 우항을 나눠보면 e^b1이 된다.
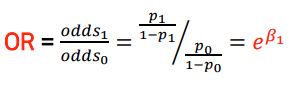
- 결과적으로 위험요소 노출여부 변수(X)의
회귀계수B1에다가 e^x지수함수를 취해주면 -> 질병에 대한 위험요소의 OR(odds ratio)다
- X=1을 대입 후, 양변에 exponentiate(e, 지수함수)을 취해주자.
- 설명변수가 1-> d개로 늘어났다.
-
설변변수 1개일 때와 똑같은 식에다가 각 설명변수 + 곱해질 회귀변수를 더해주기만 하면 된다.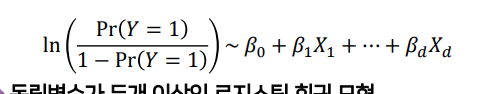
-“Adjusted” logistic regression model이라고 부르기도 한다
-
설명변수 1개일 때의
OR <-> b간의 관계가 똑같다.- 즉, Xj의 회귀계수에 지수함수 -> 결과변수에 대한 j번째 위험요소의 OR (나머지 독립변수들은 고정)

- 즉, Xj의 회귀계수에 지수함수 -> 결과변수에 대한 j번째 위험요소의 OR (나머지 독립변수들은 고정)
-
만일 X1만 위험요소 노출여부라고 보고, 나머지 X2~Xd를 confounder들이라고 가정하면
- e^b1 = 위험요소의 OR (단, 나머지 confounder 값들이 고정되어있을 때!)
- 주의점: sample size에 비해 너무 많은 독립변수들을 넣으면 안된다.
오버피팅: 내가 가진데이터의 정보량에 비해, 너무 복잡한 모델(변수가 많은 모델)을 fitting하게 됨으로서, 그 데이터의 signal뿐만 아니라데이터가 가진 noise까지 fitting에 사용되어 문제가 발생한다.- 기준은 없지만, 일반적으로
1개 독립변수추가시 마다 환자 수10~20명(데이터)가 필요하다- 즉, 데이터 10개마다 1개 변수, 20개마다 1개 변수를 사용할 수 있다.
- 예를 들어, 100명의 연구대상자를 가지고 연구한다
- 10으로 나누면 10개까지 독립변수 가능
- 20으로 나누면 5개까지 독립변수 가능
- 위험요소 X가 있을 때,
X의 효과( = 질병과의 상관관계)를OR(or RR)로 젤 수 있는데- X의 OR이, 다른 변수 A에 의해 달라질 때, X와 A간의 인터렉션이 있다고 한다.
-
X와 A간 인터렉션이 통계적으로 유의한가?도 판단해야한다.
-
A값별X의 y에 대한OR이 통계적으로 다른가?를 판단해야한다. - 로지스틱 회귀분석의 옵션 중
interaction term을 넣어서 분석하면 된다.interaction term: 두 변수 X와 A를 곱한 변수(X X A)
-
- 왜 두 변수를 곱한 것이 interaction term이라는 로지스틱 회귀분석의 추가 변수가 되는가
- 앞에서
석면과흡연간에 인터렉션이 있는가?를 본적 있다.
- 앞에서
-
X석면(X1)의 효과가, A흡연(X2)에 따라 다른가?의 인터렉션에 관심있다고 가정하고 보면- 로지스틱 모델 회귀식을 쓰되, 인터렉션 텀인
b3(X1 * X2)도 추가해서 작성하자- ln(질병O 그룹의 오즈) = b0 + b1X1(석면노출여부에 대한 term) + b2X2(흡연노출여부에 대한 term) + b3X1X2(두 변수를 곱한 인터렉션 term)
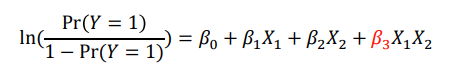
- ln(질병O 그룹의 오즈) = b0 + b1X1(석면노출여부에 대한 term) + b2X2(흡연노출여부에 대한 term) + b3X1X2(두 변수를 곱한 인터렉션 term)
- 이제 interaction을 발생시키는
A(흡연, X2)변수의 값별로 X의 OR을 봐야한다.-
X2 = 0(흡연여부가 비흡연인 경우)일 때,X1의 OR(X1 앞에 달려있는 회귀계수에 지수함수 취하면 됨)을 보기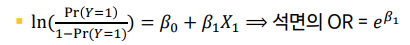
-
X2 = 1(흡연여부가 흡연인 경우)일 때,X1의 OR을 보기-
X1의 회귀계수가 인터랙션 term때문에
(b1 + b3)X1으로 바뀌게 되었다.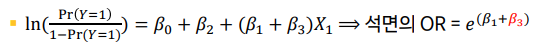
-
X1의 회귀계수가 인터랙션 term때문에
-
- 이제 A값별 구한 OR들을 비교해보자.
- 비흡연자의 석면OR
e^b1vs 흡연자의 석면ORe^(b1+b3) - 만약, b3가 0이라면, 비흡연자와 흡연자의 석면OR이 동일해진다.
- 즉, b3 = 0이라면 인터랙션이 없다라는 뜻이다.
- 비흡연자의 석면OR
- b3 = 9인지 아닌지 검정 -> 인터랙션 없는지 있는지 검정하는 것과 같다 -> 흡연여부에 따른 석면OR이 변화가 있는지 없는지 검정하는 것과 같다
- 로지스틱 모델 회귀식을 쓰되, 인터렉션 텀인
head(dat1)
# -> logistic regression으로 보기
# 로지스틱 회귀분석은 glm()메서드를 이용한다.
# - 칼럼만 입력하며, 자동완성 안되는 메서드임.
# - family에 회귀 종류 기입
# - 회기분석은 메서드 결과를 변수에 받아놓고, summary()해서 보는게 좋다.
obj1 <- glm(nasal.cancer ~ exposure.grp, data = dat1, family = "binomial")
summary(obj1)
- 제일 중요한 것은
Coefficents:의 회귀계수(코 에피션트) 부분이다.Coefficients: Estimate Std. Error z value Pr(>|z|) b0 <- (Intercept) -2.6750 0.1615 -16.567 < 2e-16 *** # 상수항 b1 <-exposure.grp1 2.0508 0.3584 5.722 1.05e-08 *** # b1의 추정치
-
glm()의 결과물 obj ->
summary()가지고는 우리가 관심있는measure of association인 e^b의 OR가 안보인다.- exp( 각 회귀계수 Eistimaated 추정치)를 넣으면 직접계산도 가능하다.
-
"broom"패키지dml tidy()를 이용하여 glm() -> obj ->tidy()로- 회귀계수(Coefficient)들 대신 e^(b)의 OR을 구할 수 있다.
library(broom)
obj1 <- glm(nasal.cancer ~ exposure.grp, data = dat1, family = "binomial")
# summary(obj1)
# tidy(obj1) # 특별한 옵션을 주지 않으면, 그냥 b의 추정치가 나온다.
# exponentiate = T 옵션을 주면, estimate(변수명)은 똑같지만, OR이 찍혀있게 된다.
# tidy(obj1, exponentiate = T)
# OR에 대한 신뢰구간(conf idential int erval)옵션까지 줄 수 있다.
tidy(obj1, exponentiate = T, conf.int = T)
- intercept의 b0의 OR은 볼 필요 없다
-
exposure.grp1(X)의 OR을 (e^회귀계수 없이) 바로 볼 수 있다.
- OR -> estimate:7.77
- p.value -> 1.051706e-08
- conf.low ~ conf.high
# confounder로 생각되는 변수(첫 노출 나이)를 + 공변량으로 넣어서
# -> OR이 아니라 adjustment OR로 계산되도록 한다.
obj2 <- glm(nasal.cancer ~ exposure.grp + age1st.grp
, data = dat1, family = "binomial")
tidy(obj2, exponentiate = T, conf.int = T)
# exposure.grp1의 OR age1st.grp1dml OR이 각각 나온다.
# 해석: age1st.grp1그룹의 값이 일정할 경우 -> exposure.grp1의 OR이 6.86으로 추정된다
# OR의 p-value가 작고 + 신뢰구간까지 제시
# R에서는 a + b + a*b 대신 -> a*b만 넣어주면, 각각의 main effect가 반영된 interaction term까지 고려 모델이 된다.
obj3 <- glm(nasal.cancer ~ exposure.grp * age1st.grp
, data = dat1, family = "binomial")
tidy(obj3, exponentiate = T, conf.int = T)
# 해석: interaction term을 의미하는 [exposure.grp1:age1st.grp1]는 OR이 중요한게 아니라 p-value가 중요하다.
# -> 3.02 x 10^-1 = 0.3 으로 유의하지가 않다.
# --> inetraction term(exposure.grp1:age1st.grp1)의 p-value가 유의하지 않으면 -> interaction이 유의하지 않다로 해석한다.
# --> 첫노출나이 그룹에 따른, exposure이 바뀌는 것이 통계적으로 유의하진 않다.
# -> 만약, interaction이 유의한 경우라고 가정한다면???
# interaction을 일으키는 첫노출나이의 0일때의 OR 과 1일때의 OR을 확인해야한다.
# -> 각 변수들의 회귀계수를 구해야 -> 각 OR을 알 수 있다.
# summary(obj3) # 회귀계수를 포함한 전체 정보가 나온다.
coef(obj3)# 회귀계수만 벡터정보로 얻을 수 있다.
# -> 단일 변수의 회귀계수로 OR을 구하면, interaction변수 0일 때의 OR임
exp(coef(obj3)[2])
# -> 단일 변수의 회귀계수 + interaction term의 회귀계수의 합 에 exp
exp( coef(obj3)[2] + coef(obj3)[4] )
실제 연구 중 confounder와 effect modifier(interactio n유발변수)가 있다고 의심이 된다?
- 일단은
confounder와 effect modifier 의심변수(A)을 무시하고, 관심있는 위험노출변수의 OR(crudeOR)만 계산한다. - A변수의 값별로 따로 따로 OR (stratum-specific OR)을 계산한다.
- A변수=0은 a*b로 만든 회귀 모델 중 자신의 회귀계수로 OR
- A변수=1은 a*b로 만든 회귀 모델 중 (자신의 회귀계수 + axb의 회귀계수)로 OR을 구했다.
- 아래와 같이 경우를 나눠 판단한다.
- Stratum-specific OR들이 비슷하고 crude OR과도 비슷할 경우
- confoundin이나 interactio이 없다
-
Stratum-specific OR들은 비슷(interaction없다)하지만 crude OR과는 차이가 클 경우
- A가
confounder이다. M-H OR(종합 OR)이나 adjusted OR(로지스틱 회귀를 이용)을 계산한다
- A가
-
Stratum-specific OR이 많이 다르다
-
interaction이 있다. 로지스틱 회귀분석을 이용하여 interaction이 있는지 검정하고 A변수 값 별 OR을 보고한다
-
- Stratum-specific OR들이 비슷하고 crude OR과도 비슷할 경우
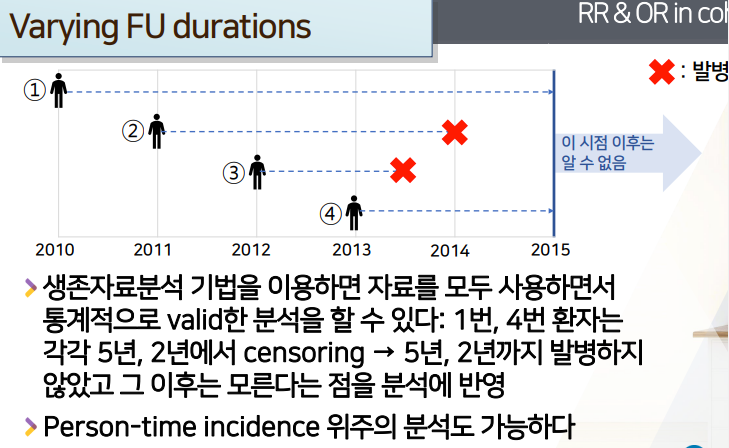
이분형의 발병여부 -> 분석시점에만 고려하면, FU기간 기준이 없어서 통계적으로 invalid
실제로 코호트 연구에서 2분형 변수가 ideal하진 않다. 그 이유는 많은 경우가 코호트 스터디 환자마다 follow up기간이 다르기 때문
- 동시에 시작해서 동시에 관찰이 끝나면 문제가 없지만
- 많은 경우, 연구 시작 -> 환자 등록은 중간 중간 이루어져서 추가됨
- 반면, 연구가 끝나는/분석하는 시점은 딱 한 시점
-
예를 들어, 1년마다 환자 등록이 각각된, 시작은 2010년 코호트 연구
-
Q. 4번 환자가, 연구시점/분석시점(2015)엔 발병안했지만, 2016에 발병했다면? 어떻게 해석해야할까?
-
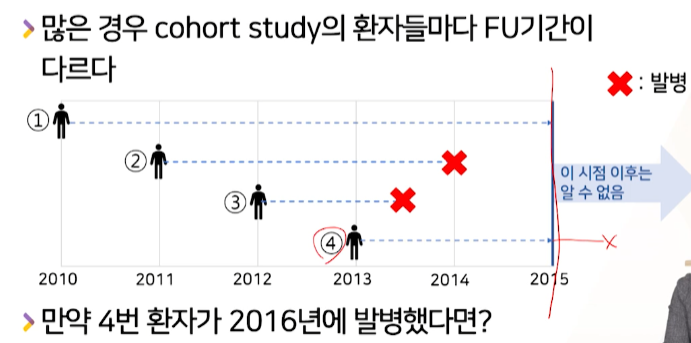
2015 시점 분석시, 2,3번 발병 / 1,4번 발병안함 -> 2016년엔 ??
- 4번이 2016년에 발병했다면?, 2번과 똑같이 3년만에 발병인데, 분석시점이후라서 카운팅 안해도 되는가?
- 공평한 분석이 아닌 것 같다.
- 공평한 분석이 아닌 것 같다.
- 4번이 2016년에 발병했다면?, 2번과 똑같이 3년만에 발병인데, 분석시점이후라서 카운팅 안해도 되는가?
-
FU(follow up) duration의 차이를 무시하고, 딱 분석시점에 잘라서 해도 되는 걸까?(코호트는 추가되는 환자등록이 다르고, 마지막 자르는 시점은 같음)
- 각 환자별 동일한 기준으로 질병발병여부를 젠 것이 아니므로, 통계적으로 valid하지 않은 것이 된다.
-
Q. 4번 환자가, 연구시점/분석시점(2015)엔 발병안했지만, 2016에 발병했다면? 어떻게 해석해야할까?
이분형의 발병여부 -> 분석시점 기준 FU기간만 제일 짧은(제일 최근 등록)환자 기준으로 바꾸면 통계적으로 valid? but FU기준을 넘어가는 정보들을 미반영
-
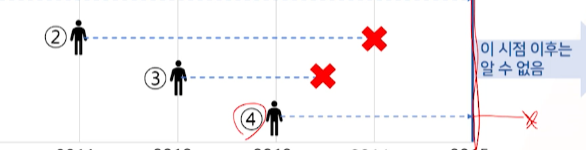
Q. 발병되서 끝난 여부와는 상관없이
FU기간이 제일 짧은(분석시점에서 제일 가깝게 등록)한 환자를 기준으로 모든 환자를 똑같이등록이후 같은 기간만 FU해서 판단한다면?- 젤 짧은 4번 환자 2년
- 1번은 2년내 발병 X
- 2번은 2년내 발병 X
- 3번은 2년내 발병 O
- 4번은 2년내 발병 X
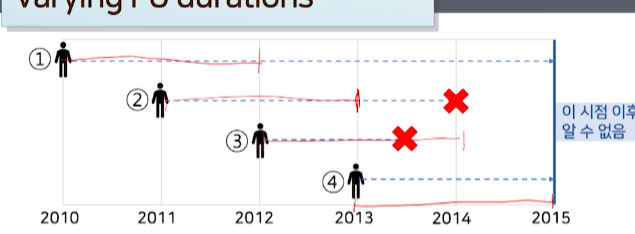
- 통계적으로는 valid하겠지만, 안타깝게도, 2번 환자가
FU기간 기준 내에선 발병X지만, 실제로 3년차에 발병O한다는 사실을 분석에 반영못하게 된다. 사실 정보를 가지고 있지만 반영 못하게 된다
- 젤 짧은 4번 환자 2년

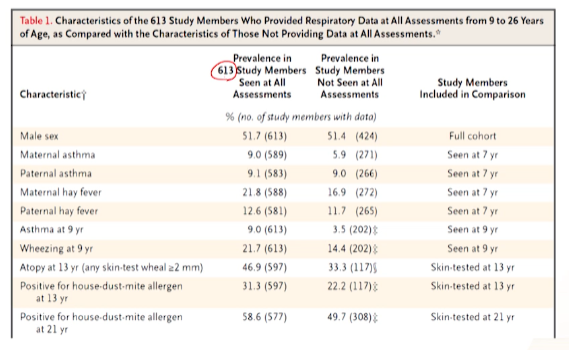
- 뉴질랜드 어느 마을에서 72년 4월 ~ 73년 3월까지 태어난 나이 1000명 중 90%가 참여
- 이후 5, 7, 9, 11, 13, 15, 18, 21, 26세에 FU
- skin-prick test: 알러지 테스트
- 폐기능 검사
- airway responsiveness : 천식관련 outcome
-
천식 발병O/X를 나누기 위해 여러가지 outcome 정의(2분형 -> 여러가지로)
- 발병이후 쭉 위징 : 펄시스턴스 위징
- 발병이후 쭉 위징있었다가 나아지면: 리미션
- 발병이후 쭉 위징 나았다가 다시 생기면: 릴랩스
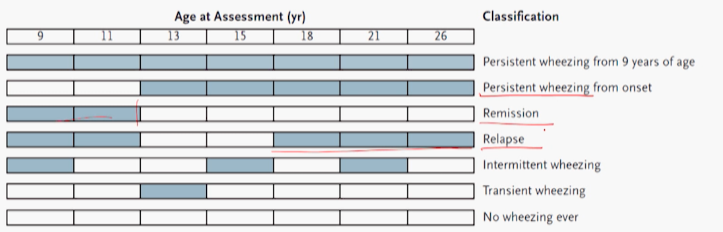
- 모든 FU에 다 참여한 아기 대상으로
- 여러가지 정의했던 outcome(이분형X)에 대해 얼마나 발생했는지 본다
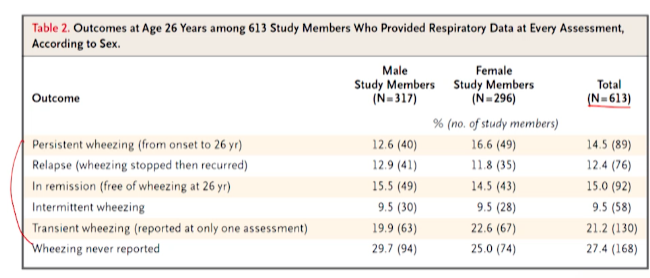
- 각 환자 특성별로 나누어 여러 outcome의 분포를 확인
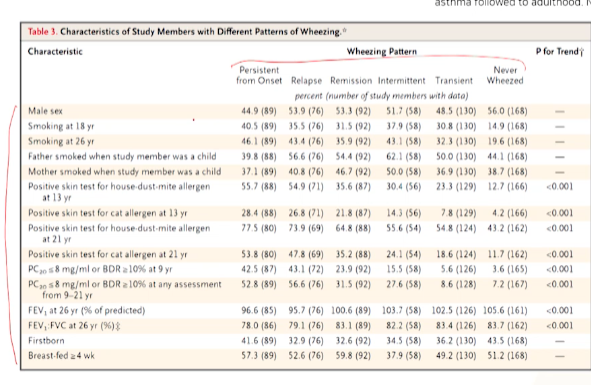
코호트 논문에서 관심있는 결과해석은 OR 분석표다
-
2분형 결과변수는 아니였지만, 2가지 outcome만 OR을 계산하게 되었음.
-
1) 위험 요소 O인 경우가 (그렇지 않은 경우에 비해)
OR이 몇 배고, 2)P value통계적으로 유의한지 보고 3) 점추정값의신뢰구간도 보자 - 4) 해석은
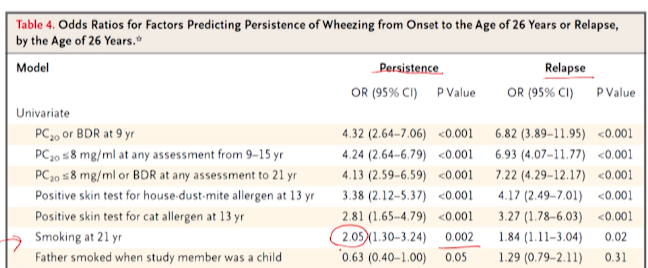
(위험요소 O)~ 한 경우가 ~하지 않는 경우에 비해outcome O될 확률이 OR 배였다- Persistence하고 Relapse에 대해 OR을 계산했는데,
-
21세에 스모킹 O인 그룹이 (그렇지 않은 그룹에 비해) Persistence가 있을 오즈가 2.05배였다
-
다변량 모델=컨파운딩 등을 보기 위해(나머지 설명변수 고정상태를 만드는 모델)OR이 유의한 설명 변수만 집어넣고 다시 모델을 만든 것.각 변수에 대해 나머지 변수는 고정일 때로 해석가능하다- (다변량모델에서, 나머지 변수들이 고정되어있는데도~) 21세 흡연여부가 -> OR이 1보다 높고 통계적으로 유의하다.
-
딱 1시점에서 데이터를 보고위험요소 노출 O/X 여부및질병 발병 O/X 여부를 보게 됨- 변화하는 관심 인구집단의 한 시점의
스냅샷으로 -
incidence(속도)를 볼 수 없고, prevalence만 보게 된다.- 위험요소가 먼저였는지, 질병이 먼저였는지 알 수 없음

- 위험요소가 먼저였는지, 질병이 먼저였는지 알 수 없음
- 변화하는 관심 인구집단의 한 시점의
- 템포럴 바이러스는
위험요소노출 여부 vs 질병여부 어떤 것이 먼저인지 알 수 없을 때오는 bias다- 예를 들어, 운동 여부 vs CHD 발병 여부
- CHD진단 받은 환자들이 운동을 늘렸을 가능성이 있다.
- 그 때 Crross-section study에 잡히면 ? 상관관계가 과소추정된다.
- 운동량 적을 수록 -> CHD 많이 발견되는 상관관계를 발견했을 지라도
- 운동량 적 -> CHD 많? or CHD 많 -> 운동량 적어짐 선후관계를 알 수가 없다

- 운동량 적 -> CHD 많? or CHD 많 -> 운동량 적어짐 선후관계를 알 수가 없다
- CHD진단 받은 환자들이 운동을 늘렸을 가능성이 있다.
- 예를 들어, 운동 여부 vs CHD 발병 여부
- 서바이벌 바이어스 / 셀렉션 바이어스 / 프리벌런스-인시턴스 바이어스 라고도 한다.
위험요소 노출후 (질병O는 물론이고) 금방 사망해버린 환자들은 연구에 포함되기가 어렵다에서 오는 바이어스다- 예를 들어, 흡연 -> 폐기종 환자에 있어서
- 흡연에 의해 폐기종 발생 환자들은 비흡연 -> 폐기종 환자들에 비해, 빨리 사망함 -> Cross-sectional study에 포함되기 힘들어진다
- 살아남아 Cross-sectional study에 포함된 폐기종 환자들의 흡연률(흡연노출여부O)는, 전체 폐기종 발병 환자의 실제 흡연률보다 낮아지게 됨 -> 흡연과 폐기종의 상관관계를 과소추정하게 된다.

- bias를 많이 가질 수 밖에 없는 study이므로, Cross-sectional Study의 결과는 다른 연구 시간의 선후관계를 가진 파악 다른 연구로 가기 전의 전 단계로서, 살펴볼 가치가 있는 가설인지 확인용으로 쓴다.
- 크로스-섹셔널 연구에서는 incidence 계산 불가 -> risk 계산 불가 -> Relative Ratio의 RR대신
Prevalence Ratio로 부른다. - OR에 들어가는 p1, p0가 risk가 아닌 prevalence ->
prevalence OR로 부른다.
- Cohort study는 연구자가 위험요소에 노출된 그룹과 노출되지 않은 그룹을 일정기간 동안 추적관찰하여, 두 그룹 간 질병 발생률 (incidence)이나 질병에 의한 사망률 등을 비교하는 연구이다
- Cohort study에서는 RR과 OR을 추정하고 신뢰구간을 계산할 수 있다
- Confounding이 있을 경우 Mantel-Haenszel 방법으로 OR을 계산하거나 로지스틱 회귀분석으로 Adjusted OR을 계산할 수 있다
- Interaction이 있을 경우 로지스틱 회귀분석으로 interaction의 유의성을 검정하고 stratum-specific OR을 계산할 수 있다
- Cross-section study에서는 prevalence ratio와 prevalence OR을 계산할 수 있다
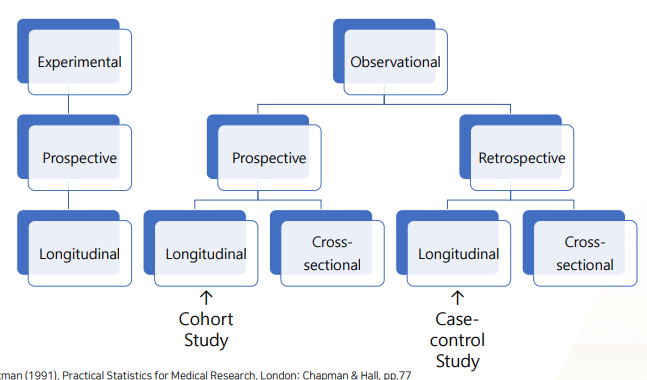
- 관찰연구의 일종이다.
- 연구는 실험연구 vs 관찰연구로 나뉘는데 관찰만 한다.
- cf) 실험연구 아니면 관찰연구 -> 현재시점 기준으로 이후(전향적) 아니면 이전(후향적) 관찰 -> 이후든 이전이든 1시점만(cross-sectional) 보냐 vs 2시점 이상(Longitudinal-이후Cohort/이전Case-control)보냐
- 연구는 실험연구 vs 관찰연구로 나뉘는데 관찰만 한다.
- 여러개의 질병을 동시에 연구할 수 없다.
- 위험요소 -> 질병발생의 선후가 정해져 있어서 질병발생 시점이 명확 -> incidence(질병발생 속도와 관련) 추정이 가능
- 위험요소 -> 질병발생의 선후가 정해져 있어서 인과관계 추론에 결정적 단서
- 위험요소 -> 질병발생의 선후가 정해져 있어서 위험요소 노출O/X이후 나올 수 있는 여러 질병들을 동시에 연구 가능하다.(타겟질병말고도 다른 질병이 나타나면 연구가능)
-
희귀병은 연구하기 어렵다.
- 코호트는
위험요소 -> 질병발생의 선후가 정해져 있어서 희귀병은 기다려도 몇 명 발생안하므로, 많은 수의 연구대상자가 확보되어야하고, 계속 follow up해야한다 - 대안: retrospective cohort study
-
연구시작 시점 이후발병하는 질병을 follow 하는 것은 동일하나, -
연구시작 시점 이전위험요소 노출O/X를 <기록(데이터),기억(인터뷰)>를 통해 결정하는 것- 질병발생률은 prospective / 위험요소 노출여부는 retrospective로서 2가지가 섞여있는 디자인이라고도 볼 수 있다.
- 위험요소 노출O/X를 기다렸다가 나누었다가 할 필요가 없이 바로 질병발생률만 관찰하므로 전체적인 연구기간이 짧아진다.
-
위험요소 -> 질병발생의 선후가 정해져 있어서 질병 발생할때까지 계속 기다리다가 위험요소 노출여부가 중간에 바뀌어버릴 수 있다.- 예> 흡연으로 위험요소 노출O였다가, 연구 중간에 금연해서 not/un exposed group으로 가버릴 수 있다
- 대안: 위험요소 노출여부를 여러번 측정 -> longitudinal study(여러번 측정시)
-
- 코호트는
-
위험요소 노출부터 질병 발병까지의 기간이 너무 긴 질병은 연구하기 어렵다.
- 정답 : 2
- 해설 : Cohort study에서는 여러개의 질병을 동시에 연구하는 것이 가능하다.
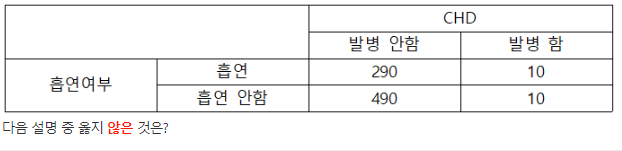
- RR(relative risk) = (10/300)/(10/500) = 1.67
- OR(odds ratio) = (290×10)/(490×10) = 0.59
- 이 데이터에서는 흡연자의 CHD 발병 위험이 더 높다.
- 흡연 여부와 CHD 발병 여부가 독립인지 검정하려면 카이제곱 검정을 시행하면 된다.
- row -> col 순으로 보고 위험요소2개 =
interaction문제인지 / 위험요소1 질병여부1의상관관계 문제인지 확인한다- 위험요소노출O/X -> 질병여부O/X 의
상관관계는위험요소O/X에 따라 group을 나눈 -> RR/OR로 판단한다.
- 위험요소노출O/X -> 질병여부O/X 의
-
상관관계를 위한 RR, OR계산은
위험요소노출 O/X group별로 나눈 질병 risk인p1/p0를 계산한다- p1(위험요소 노출O risk) = 10/10+290
- p0(위험요소 노출O risk) = 10/10+490
- rr = p1/p0 = 500/300 = 1.67
-
OR계산은
위험요소노출 O/X group별로 나눈 질병 odds(risk / 1-risk)를 구한뒤 비율을 구해야하지만위험요소-질병 표에서는a(oo)d(xx) / bc로 계산해버리면 된다.- a(oo) = 흡연->발병 = 10
- d(xx) = 흡연x->발병x = 490
- bc = 반대 대각선 = 10 x 290
- 추가적으로 범주(위험요소 노출O/X)형 범주형(질병O/X) -> 독립성 검정으로 상관관계 없음을 검정할 수 도 있다.(카이제곱 검정)
- 정답 : 2
- 해설 : 위의 표는 disease positive가 왼쪽이 아닌 오른쪽 열에 배치되어있다. 이를 일반적인 형태(disease positive가 왼쪽 열, exposed가 위쪽 행)로 바꾼 후 cross-product 공식으로 OR을 구하면 (10×490)/(10×290) = 1.69이다.