의연방06) 환자대조군 연구
Case-control Study
📜 제목으로 보기✏마지막 댓글로
- 학습개요
- Case-control study
- 정리하기
- R 고급 실습(matched case-control study with dplyr-pipeline, broom-tidy, tableone, survival-clogit) 포함됨
- Observatinal: 익스페리멘탈이 아닌 옵져베이셔널 스터디(관찰연구) 중에서
- Retrospective: 후향적 = 현재 시점에서부터 과거의 기록들을 이용하며
- Logintidinal: 적어도 2시점 이상을 관찰한다.
- Retrospective: 후향적 = 현재 시점에서부터 과거의 기록들을 이용하며
-
구체적으로 보면
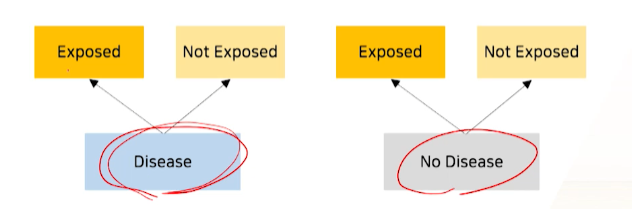
- case:
질병을 가진환자군(=case) 먼저설정 - control:
질병이 없으면서&&환자군(case)과 비슷한 대조군을 그 후설정 - 각 질병여부O/X(case/control) 그룹별 ->
과거에 위험요소노출 O/X여부(특성O/X여부)를 확인 및 비교한다-
cohort와는 반대로 순서로 순서가 정해져있음
-
cohort와는 반대로 순서로 순서가 정해져있음
- case:
-
위험요소와 질병의 상관관계가 있다면?- 코호트(위험요소 그룹별 -> 질병여부 risk, RR, OR)와 달리 질병여부 -> 위험요소 순으로 계산을 해야한다.
-
상관관계가 있다면,
질병O(case) 그룹 중 위험요소 노출O사람이질병X(control)그룹 중 위험요소 노출O보다 많을 것이다.가 기본 개념이다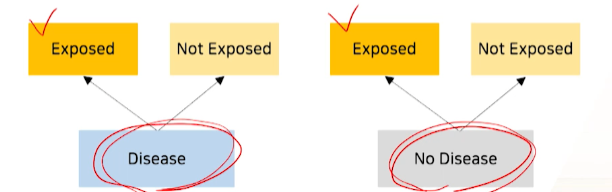
- cohort study에서는
현재 시점에 위험요소 노출여부가 먼저 결정이 되고 -> 그 이후에 질병이 생기냐/안생기냐를 본다.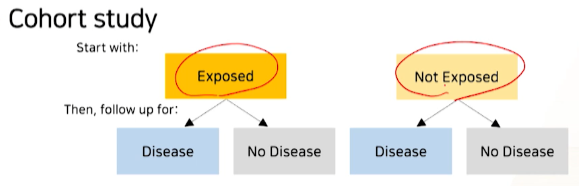
- case-control에서는
현재 시점에서 질병O인 사람/질병X인 사람을 찾은 뒤->과거에 위험요소에 노출이 되었냐/안되었냐를 본다.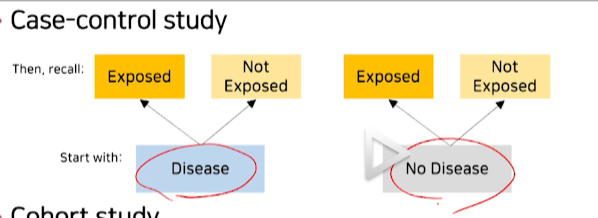
- 저렴하고 빠르다
-
cohort처럼 질병이 일어날 때까지 계속 FU하면서 기다림, 이 필요가 없다
- 지금 벌써 질병이 발생한 사람으로 시작 -> 빠르게 연구 수행 가능
- rare disease 연구에 적합하다
- 지금 벌써 질병이 발생한 사람으로 시작 -> 희귀병도 발생할때까지 기다릴 필요 X
- 발병에 오래 걸리는 질병 연구에 적합하다
- 지금 벌써 질병이 발생한 사람으로 시작 -> 발병 오래걸리는 병을 기다릴 필요 X
-
cohort처럼 질병이 일어날 때까지 계속 FU하면서 기다림, 이 필요가 없다
-
여러개의 위험요소를 동시에 연구할 수 있다
- 질병O/X가 먼저고 -> 뒤에 결정되는 (그 이전 기록을 찾아보다가) 여러개 위험요소를 동시에 연구 가능
- 대신 먼저 지정되는
질병에 대해서는 1개만 관찰 가능
- 대신 먼저 지정되는
- 질병O/X가 먼저고 -> 뒤에 결정되는 (그 이전 기록을 찾아보다가) 여러개 위험요소를 동시에 연구 가능
-
보통 cohort study에 비해 적은 수의 연구대상자가 필요하다
- 코호트는 많은 환자들을 대상으로 시작해야 -> 일부만 질병이 발병함
- 케이스-컨트롤은 애초에 발병된 사람을 잔뜩 확보한 상태로 시작 -> 그나마 적은 수로 가능
-
질병에 대해 알려진 것이 없을 때,위험요소와의 상관관계를 제안하는 초기 단계의 연구로 적합하다- 위험요소-질병의 상관관계를 알아보고 싶다면, 시간이 오래걸리는 전향적 코호트 연구 이전에,
먼저 질병O인 사람들 먼저 모아놓고 시작하는 케이스-컨트롤 연구부터 시작한다 - case-control study에서 상관관계가 있는 것 같다 -> 다음 단계로 cohort study를 비용+시간 좀 들더라도 진행하는 것이 일반적이다
- 위험요소-질병의 상관관계를 알아보고 싶다면, 시간이 오래걸리는 전향적 코호트 연구 이전에,
- 위험요소 노출과 질병의 선후관계가 불분명할 때가 많다
- (선후관계가 불분명하니)인과관계 성립이 어려움
- Case-control study에서 얻은 데이터에서는 case와 control의 비율이 연구자에 의해 인위적으로 조정되므로 prevalence에 대한 추론을 할 수 없다
- case를 먼저 몇명 모은 뒤 -> control을 계획해서 수가 정해진다. -> case:control의 비율이 연구자에 의해 인위적으로 정해진다
- 질병에 대한 prevalence(발생확률), risk를 추정할 수 없다
- bias가 생길 가능성이 아주 높다
- bias를 피하기 위해 신중하게 선택해야한다.
- Case 선택에서 오는 bias:
- case중 살아남은 사람만 연구에 포함된다. 즉, 발병 후 빠르게 사망한 환자는 연구에 포함되기 어렵다
- 정말 심각하여 발병후 사망한 환자들은 case를 선택할 때 포함되질 않는다. ->
살아남은 사람만 포함되는 bias
- 정말 심각하여 발병후 사망한 환자들은 case를 선택할 때 포함되질 않는다. ->
-
위험요소와 질병 발병(incidence)의 연관성보다는 위험요소와 ‘발병 후 생존’의 연관성을 관측하게 된다
- case를 먼저 선택하는 경우,
질병 발생 && 발병후 생존까지 포함된 개념을 ->위험요소노출과의 상관관계를 조사하게 된다.
- case를 먼저 선택하는 경우,
- case중 살아남은 사람만 연구에 포함된다. 즉, 발병 후 빠르게 사망한 환자는 연구에 포함되기 어렵다
-
Control 선택에서 오는 bias:
-
최대한 case와 비슷한 사람을 골라야 하는가 vs.최대한 control 전체를 대표(위험요소 노출 가능성이 관심인구와 비슷한)하는 사람을 골라야 하는가가 control 선택의 관건이다. -
예제) Pearl (1929): 결핵이 암을 예방하는가?
- cases : 존스홉킨스병원의 암 사망자(질병O)
- controls :
같은 기간 같은 병원의 암환자가 아니었던 사망자(질병X)- case와 비슷한 controls를 고른 경우에 해당함
-
case의 6.6%, control의16.3%가 부검에서 결핵이 발견됨
- 위험요소 노출여부 O/X를 확인함.
-
controls(질병X)에서 -> 결핵(위험요소 노출여부 X)가 많이 발견됨
- 결핵(위험요소노출X) -> controls(질병X, 암X)와 상관관계가 있다(결핵이 암예방에 효과있다)고 결론내릴 수 있다.?!
-
당시 결핵이 주요 입원요인이어서, control 그룹의 결핵 prevalence가 general population에 비해 높게 나타남
- case와 비슷한 상황을 맞추다 보니(같은기간, 같은병원), control 전체를 대표하질 못했다.
- Carlson & Bell (1929): control을 같은 기간 같은 병원의
(암X사망자) -> 심장병 사망자로 설정하여 재분석, 결핵의 비율이 case와 control에서 비슷하게 나타남-
my) case와 비슷하게 맞추면서 (같은기간&같은병원),
위험요소 노출 가능성이, 관심인구(전체인구)와 비슷해지도록, general population을 좁히기 위해, 암X사망자 -> 특정 질환 사망자로 특정해주기- 병원내 암X 사망자의 결핵 -> general population(병원 밖... 포함)의 결핵과 너무 차이남
- 병원내 & 심장병사망자의 결핵 -> general population자체가 병원내 심장사망자? 중 결핵?
-
my) case와 비슷하게 맞추면서 (같은기간&같은병원),
-
Control을 선정할 때 지켜야할 원칙
- 되도록이면, case와 같은 general population(같은 국가 or 같은 도시or 같은 병원)에서 선정한다
- 앞의 연구
-
위험요소 노출 가능성(=질병의 노출이라면 prevalence)이 general population과 비슷한 그룹으로 선정한다
- 앞의 연구에서는, general population의 위험요소(결핵) prevalence보다, controls의 prevalence가 훨시 높았다.
- 관심 질병이 발병할 가능성이 있었으나 발병하지 않은 사람으로 선정한다
- 예) 자궁암을 연구하면서 남성이나 자궁 적출한 여성을 control로 선정하면 안됨
- 과거에 발병했다가 치료된 사람은 되도록 control에서 제외한다
- Case와 control간(질병O/X 그룹별)의 위험요소 노출
비율의 차이를 관측했을 경우, 이것은 위험요소와 질병간의 상관관계를 의미한다 - 이 상관관계는 인과관계인가, confounding에 의한 것인가? 상관관계 확인될 시, 항상 confounding여부를 확인한다
- 예) 환자군이 대조군에 비해 흡연률이 높지만
-
my) 일단 case/control 그룹간 -> 위험요소 노출
비율 차이발생 ==상관관계가 확인된 상태 -> confounding 확인해야 함
-
my) 일단 case/control 그룹간 -> 위험요소 노출
- 예) 환자군이 대조군에 비해 흡연률이 높지만 소득이 낮을 경우,
- my) 위험요소 노출 이외에
소득이라는 confounding이 있을 경우 확인해야한다.
- my) 위험요소 노출 이외에
- 예) 환자군이 대조군에 비해 흡연률이 높지만 소득이 낮을 경우, 흡연이 이 질병의 위험을 높이는가, 아니면 저소득으로 인한 다른 요인이 질병의 위험을 높인 것인가?
- 저소득 -> 흡연이외에 다른 요인들이 질병 위험을 높이는지 vs 흡연에 의해 질병의 발병이 높아지는 것인지 구분해야한다.
- confounding(confounder에 의한 효과)를 없애야한다?!
- 예) 환자군이 대조군에 비해 흡연률이 높지만
- Confounding(에 의한 효과)을 없애기 위해,
confounder로 의심되는 요인의 분포가 환자군과 대조군에서비슷해지도록matching을 수행할 수 있다- Matched case-control study
- my) Matched = confounding(효과)를 제거하기 위해 confounder분포를 case-control 그룹별로 맞춰놓은 것
- 주의 : 모든 case-control study가 matching을 하는 것은 아님
- 항상 그런 것은 아니다! 해석 방법이 다르다
- Matched case-control study
- group matching
- case를 먼저 선정한 후 -> case에서 매칭변수(=confounder로 의심되는 변수)의 분포를 알아보고 -> 동일한 분포를 가지는 control을 선정한다
- my) case 뽑고난 뒤, confounder를 고려해서 control을 뽑는다는 얘기
- 실제론 자주 안쓰임
- 예) case에서 저소득이 30%일 경우, control도 저소득이 30%가 되도록 선정
- case를 먼저 선정한 후 -> case에서 매칭변수(=confounder로 의심되는 변수)의 분포를 알아보고 -> 동일한 분포를 가지는 control을 선정한다
- individual matching (matched pairs):
- 실제로 자주 쓰임
- case군 먼저 선정 -> 각각의 case 환자 1당 매칭변수 값이 비슷한 control(1명)을 찾아 짝을 짓는다
- my) 한명씩 confounder를 고려해서 짝지어서 선발
- 예) 1번 case의 소득과 비슷한 소득을 가진 1번 control을 선정, 2번 case의 소득과 비슷한 2번 control을 선정...
- 같은 동네에 사는 주민, 형제 자매, best friend 등을 control로 선정하기도 한다
- 1명, 1명이 아니더라도, 여러가지가 비슷한 group이면 한번에 뽑기도 한다.
- 매칭변수가 많으면 매칭이 어려움 -( 20개가 confounder면...) 비슷한 control을 찾을 수가 없으므로
- 매칭변수의 효과는 분석 불가
- case와 control간 매칭변수의 분포가 같아지므로 (같아놓게만 만들어놓고, 분석에는 사용 안됨)
- 예를들어, 나이를 매칭변수(=confounder)로 지정했다면, 나이분포는 서로 비슷하다 -> 질병여부에 독립이 되어버림 -> 질병과의 관계를 분석 못하게 됨
- case와 control간 매칭변수의 분포가 같아지므로 (같아놓게만 만들어놓고, 분석에는 사용 안됨)
- overmatching (unplanned matching)
-
매칭변수를 지정했는데, 관계있던 변수들까지도 의도하지 않게 매칭된 경우를 말함.
- ex> 소득을 매칭변수로 사용했는데, 소득과 비슷한 변수(생활습관, 식생활)까지도 비슷해져버려서 분석이 안됨
- 예) 소득에 대해서 매칭하였더니 생활습관, 식생활 등 다른 변수들까지 비슷해져서 관심 위험요소 분포까지 비슷해짐
- 또는, control을 비슷한 best-friend로 지정했는데, case와 비슷한게 너무 많아, 위험요소까지도 의도하지 않게 매칭된 경우를 말함.
- 예) 경구용 피임약과 자궁암의 관계 연구를 위해 best-friend control을 선정 : 친구끼리는 경구용 피임약 사용여부가 거의 같아서 환자군 대조군 간 경구용 피임약 사용여부가 의도하지 않게 매칭이 됨
-
매칭변수를 지정했는데, 관계있던 변수들까지도 의도하지 않게 매칭된 경우를 말함.
-
전체 연구대상자 수가 고정되어 있는 경우는 controls:cases = 1:1일 때 가장 파워가 높다
- 파워 높다 = 관측하고자 하는 차이를 detect할 가능성이 가장 높다
-
Case-control 스터디는 대부분의 경우 case의 수가 고정되어 있고 vs control은 충분히 많다
- 2:1, 3:1, 4:1까지는 대조군 수를 늘릴 수록 파워가 눈에 띄게 증가한다
- 4:1 이후(5:1부터)로는 파워 증가가 미미하다
- 적은 case 1명당 ->
control은 4명까지만 모아라
- 적은 case 1명당 ->
- Case-control study에서는 RR을 직접 추정할 수 없다. OR은 추정 가능하다
- rare-disease의 경우, OR≈RR: 비슷하기 때문에 OR 구하고 -> RR도 그럴 것이다.
- case-control study에서는
모집단에서 연구자가 질병O / X의 그룹별n수를 직접 선택해서...- 질병의 prevalence(발생 확률)이 인위적으로 설정되어 -> risk도 인위적인 값일 수 밖에 없다
- 모집단에서의 risk(아직 case 선택 전)
- 모집단에서는 risk를 계산할 수 있다. ( 위험요소노출여부 그룹별 -> risk 계산 -> risk비율 계산)
- 폐암인 사람 100명 / 폐암 아닌 사람 5000명 / RR = 4
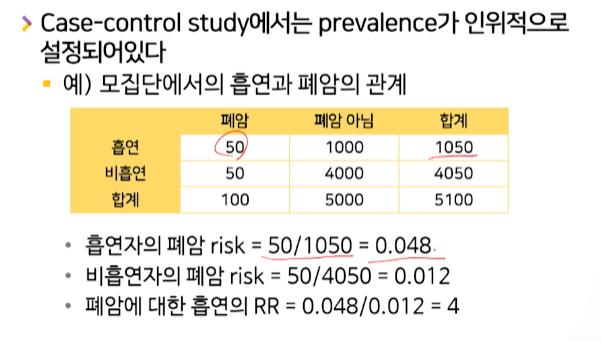
-
연구자가 뽑은 case -> control
- 폐암환자 100명은 모두 다 사용하였다
-
하지만 ,폐암 아닌 사람은 5000명 중 100명만 뽑아서
controls:case = 1:1로 표집하였다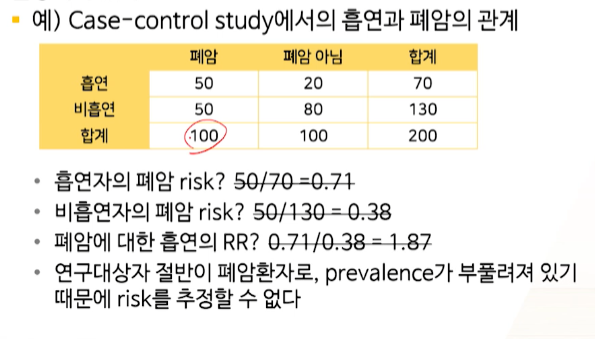
-
controls는 100명으로 줄었지만, 위험요소 노출O/X 비율 1:4는 그대로 유지하여 잘 뽑혔다.

- 이제 case-control study에서 risk를 계산해보자.
- 흡연O 폐암risk: 50/50+
20-> 0.71- 흡연한 사람은 폐암발생확률이 71%다 -> 아무도 흡연 안할 것. 너무 높은 위험
- 흡연X 폐암risk: 50/50+
80-> 0.38- 흡연을 하지 않아도, 폐암발생확률이 38% -> 끔찍한 상황의 높은 위험
-
controls를 case와 1:1로 표집 ->
controls의 n수를 굉장이 낮춰 case의 prevalence가 뻥튀기 -> risk도 뻥튀기된 상황이기 때문 -> risk 추정 불가능 -> RR 추정 불가능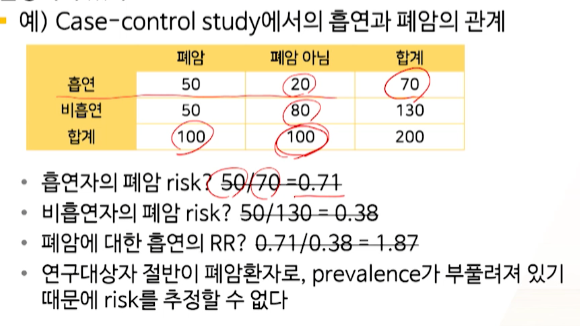
- 흡연O 폐암risk: 50/50+
-
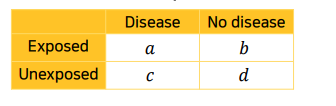
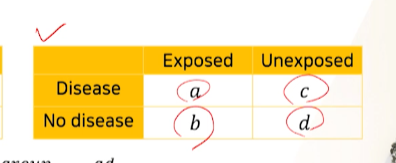
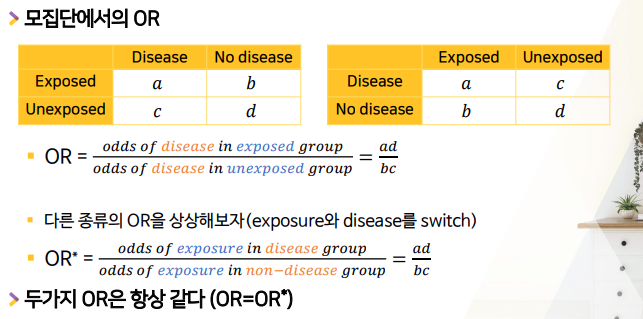
모집단 OR = a(oo)d(xx) / bc

-
case-control에서의 OR (표 반전 후, ad/bc)
-
a와 d는 그대로 있고, b와 c만 위치 바꼈다 -> 공식에는 동일하게 ad/bc
-
- Case-control study에서는 controls(질병X)를 줄여서 1:1~4:1정도만 표집하느라 상대적으로 case그룹(질병O그룹)이 뻥튀기된 상태다
- 실제 모집단에서의 유병률(prevalence 질병/전체)보다 훨씬 높아진다.
- 아래 질병O 컬럼의 숫자들이 실제보다 비율상 커져있게 된다.
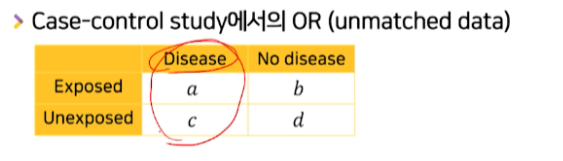
- risk의 계산 방법인 (위험 요소 노출O row / 위험요소 노출X row)별 -> 가로별 risk(질o/질o+질x)가 의미가 없어진다.
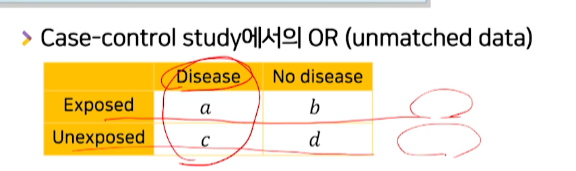
- 질병O 칼럼 뻥튀기 -> risk 뻥튀기로 p 계산 불가 -> RR 및 OR(p/1-p 비율) 계산 불가
- 처럼 보이지만, OR은 ad/bc로 계속 사용할 수있다. 분모분자속 a와 c가 똑같은 비율로 뻥튀기 되어있기 때문이다.

- 처럼 보이지만, OR은 ad/bc로 계속 사용할 수있다. 분모분자속 a와 c가 똑같은 비율로 뻥튀기 되어있기 때문이다.
- RR은 직접 추정할 수 없으나 rare disease(레어 디지즈)인 경우에 한해서는 OR과 RR 값이 비슷하다
- 신뢰구간 및 위험요소-질병 상관관계(독립성)검정은 카이제곱 검정 -> cohort study와 동일하다
- confounding과 interaction을 다루기 위해 로지스틱 회귀분석 이용 -> cohort study와 동일하다
-
로지스틱 회귀분석을 이용한 measure 추정은- cohort study -> 연습했던 OR외 risk, odds 추정도 다 가능
- Case-control study -> OR만 추정 가능
- 일단 matching된 환자들끼리는 ->
서로 독립이 아니다를 분석에 반영해야한다.- (독립이 아니면)
OR 공식이 더이상 성립X -
(독립이 아니면)
위험요소 - 질병 상관관계(독립성) 검정을 카이제곱으로 더시아 X- 1:1매칭일 때 : McNemar’s test
- c:1매칭일 때 : Cochran-Mantel-Haenszel test
3.실제 Adjusted OR 계산, interaction 분석 :
conditional logistic regression
- (독립이 아니면)
- 기존 데이터 구조
- 위험요소 노출(E)요부 row -> 질병 여부(D) col
- 위험요소 노출(E)요부 row -> 질병 여부(D) col
-
1:1로 individual matching한 경우 데이터 구조
- 데이터 구조가 바뀌는 이유는, 상관관계의 정보가 없는 = 결과의 차이가 없는 n수를 한눈에 처리하기 위함이다?!
- matching되어 짝이 정해진 상황이며 && 특히 1:1이라면, 1명은 control 1명은 case이므로
- my) 칼럼쪽에 있던 질병O/질병X를 row/col으로 나누되, 층을 하나 더줘서 나눴다?!
- my) 칼럼쪽에 있던 질병O/질병X를 row/col으로 나누되, 층을 하나 더줘서 나눴다?!
- 짝지어진 pair내에서
- 둘다 exposed O -> n11
- 둘다 Unexposed -> n22
- case만 exposed O -> n12
- controls만 exposed O -> n21
- 선후관계에서 뒤쪽인 exposed여부에 따라 n을 세어주는 sense
- 각 자리에 몇개의 matched된 pair가 들어갔는지 확인할 수 있다.
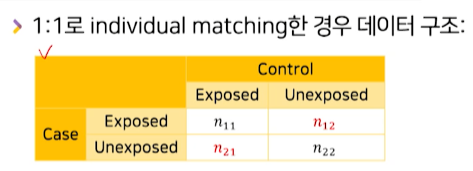
- n11, n22는 매칭된 쌍들 중, 둘 다 exposed됬거나, 둘다 unexposed 된 것이므로
선후관계에서 결과 뒤쪽인 외험요소 노출 차이를 만들지 못하기 때문에상관관계에 대한 정보가 없는 카운팅들이다.
-
case와 control간에 위험요소노출여부가 다른 cell만 상관관계에 대한 정보를 준다
- n12 ->
case는 위험요소노출 O/ control은 X -> n12는 많을 수록, 위험요소는발병위험을 높인다. - n21 -> case는 X /
control은 위험요소 O-> n21는 많을 수록, 위험요소는발병위험을 높이진 않는다.
- n12 ->
-
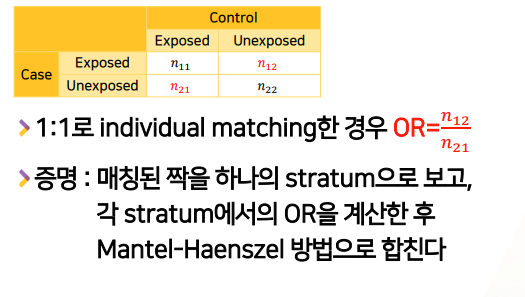
matched Case-Control study에서 OR 계산법
- OR = n12/n21 = CaseExposed(O)&ControlExposed(X) / CaseExposed(X)&ControlExposed(O)

- 증명
- OR = n12/n21 = CaseExposed(O)&ControlExposed(X) / CaseExposed(X)&ControlExposed(O)
-
이전
- confounder가 있을 때, confounder의 값 별로 stratum을 나누고
- 각 stratum(스트레이 텀)마다 OR을 구한뒤 M-H방법으로 합쳤었다.
-
여기선
-
각 matcing pair를 stratum으로 보고, 매칭 쌍마다 OR을 구한 뒤, 합친다
- 매칭된 쌍이 총 I개 있는 경우

- 매칭된 쌍이 총 I개 있는 경우
-
matched case-control용 데이터구조에서 1cell( n ) 마다 -> cohort나 unmathced 의 2by2 table로 변경할 수 있다.???- 예를 들어, n11(case-exposed & control-exposed) 이라면 ->
- case-exposed :ai = 1
- control-exposed :bi = 1
- 나머지 ci, di = 0
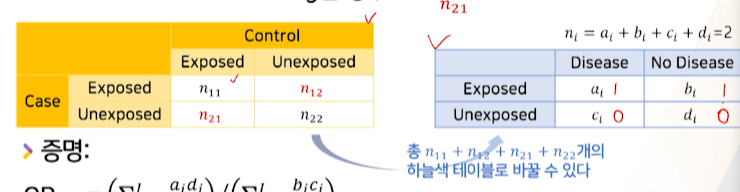
- 예를 들어, n11(case-exposed & control-exposed) 이라면 ->
-
다하면 아래와 같다고 한다.

-
각 matcing pair를 stratum으로 보고, 매칭 쌍마다 OR을 구한 뒤, 합친다
-
my) matched case-control study에서 OR은
- 데이터 구조가 조금 다르다(위층row-case, col-control 상태에서 아래층에는 위험요소노출여부)
- OR계산은 n12/n21 = case-exposed & control-unexposed / case-unexposed & control-exposed
- my) 앞1:case-exposed / 뒤2:control-unexposed
- my) 앞2:case-unexposed / 뒤1:control-exposed
- 기존 cohort, unmatched case-control study에서는
카이제곱 검정- 범주형 변수 2개가 서로 독립인지 확인
- matched case-control study
- 범주형 중 2분형 변수 2개가 있고, 서로 짝지어진 matched 데이터일 때, 연관성 검정 ->
멕니마 검정
- 범주형 중 2분형 변수 2개가 있고, 서로 짝지어진 matched 데이터일 때, 연관성 검정 ->
- McNemar's test
- H0 : 환자군과 대조군 간에 위험요소 노출비율의
차이가 없다⇔ 𝑛+1 = 𝑛1+ ⇔ 𝑛21 = 𝑛12- case환자군 위험요소 노출 total = n1+
- control대조군 위험요소 노출 total = n+1
-
2개가 같아야한다.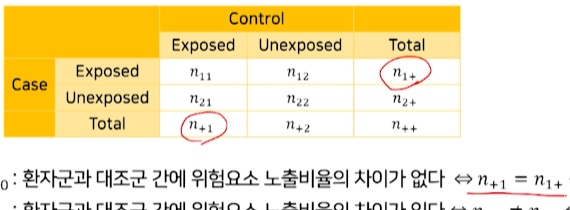
- n1+ = n11 + n12 / n+1 = n11 + n21 이므로 2개가 같다면
n12 = n21이 같다가 귀무가설
-
- H1 : 환자군과 대조군 간에 위험요소 노출비율의 차이가 있다 ⇔ 𝑛+1 ≠ 𝑛1+ ⇔ 𝑛21 ≠ 𝑛12
- 반대로
n12 = n21이 같지 않다가 대립가설
- 반대로
- 검정통계량 : 귀무가설 아래에서 자유도 1인 카이제곱 분포를 따른다
-
결국에는 n12와 n21의 차이가 얼마나 크냐를 보게 되는 것이며, 분포는 카이제곱 분포를 따른다 -> OR이 1이냐 아니냐로 차이가 얼마가 나는지 본다.

-
결국에는 n12와 n21의 차이가 얼마나 크냐를 보게 되는 것이며, 분포는 카이제곱 분포를 따른다 -> OR이 1이냐 아니냐로 차이가 얼마가 나는지 본다.
- H0 : 환자군과 대조군 간에 위험요소 노출비율의
- Conditional logistic regression 1개로 OR 종합 및 검정을 퉁쳐버린다.
- 모든 것을 한번에 해결(OR 추정, OR이 1인지 아닌지, OR의 신뢰구간)
- standard logistic regression에서 딱 1가지만 다르다.
- 매칭을 분석에 반영해야함.
- 매칭된 쌍마다 고유한 상수를 b0대신 ai(상수, intercept)를 넣어준다.
- 매칭 = 비슷한 경향 = 질병 발생 확률 비슷 = Odds에 로그 취한 것도 비슷 = 비슷하게 만들어주기 위해 상수 더해줌
- ai값 자체는 중요하지 않음.(추정하지 않음) 관심있는 회귀계수만 결국 계산됨.



- 연구질문: 인위적으로 임신중절을 했을 경우,
secondary infertility에 영향을 미치는가 - case/control 선택
- case : secondary infertility
- control : 나이, 출산자녀 수, 교육수준으로 매치된 건강한 여성
- 2:1로 매치됨 (83 cases, 83×2-1=165 controls)
- 1명의 case는 control 1명 밖에 못찾아서 .. -1
head(infert)
dat.i <- infert
summary(dat.i)
# parity: 자녀 수, induced: 중절 횟수, case: secondary infertility여부
# spontaneous: 자연유산 횟수 / stratum: 누가 누구랑 매칭되어있는지를 보기 위한 id(84번부터 control에 해당하며 id1이랑 매칭되어 1)
# 관심있는 칼럼: case <- induced
library(dplyr)
dat.i2 <- dat.i %>%
mutate(induce.ever = ifelse( induced > 0, 1, 0))
head(dat.i2)
dat.i2 <- dat.i %>%
mutate(induce.ever = ifelse( induced > 0, 1, 0),
spontaneous.ever = ifelse( spontaneous > 0, 1, 0))
head(dat.i2)
dat.i2 <- dat.i %>%
mutate(induce.ever = ifelse( induced > 0, 1, 0),
spontaneous.ever = ifelse( spontaneous > 0, 1, 0)) %>%
mutate_at(vars(induced, induce.ever,
spontaneous, spontaneous.ever),
list(~as.factor(.)))
head(dat.i2)
summary(dat.i2)
# as.factor() 로 범주형 변수로 만들면, summary시 counting이 잘된다.
# -> case와 control에 대한 전반적인 비교를 해야한다.
# 4-1. descript하게 보기 위해 table을 하나 만들어준다.
# -> tableone 라이브러리를 이용함
library(tableone)
tobj <- CreateTableOne(vars = c("age", "parity", "education",
"induced", "spontaneous"), # 2) strata에 넣은 그룹(case vs control)별로, 보고 싶은 데이터 칼럼
strata = "case", # 1) case와 control을 범주종류로 나눈 칼럼
data = dat.i2)
tobj
print(tobj, test=F) # p-value를 제외하고 보는 방법: print( , test=F)
# 해석
# - 이미 match를 age/aprity/education기준으로 맺어놓은데이터다 -> case와 control에서 분포가 비슷할 것이다.
# - induced는 control과 case가 분포가 비슷하다
# - spontaneous는 control과 case가 분포 차이가 난다.
# -> 특히, 자연임신중절이 0은, control일 때 훨씬더 많다.
# --> 우리의 가설: 인공임신중절(induced)와 case/control여부와 관계있을 것 같았지만,별로 관계없어보이고
# --> 오히려 자연임신중절(spontaneous)과 관련있어 보인다.
# --> 그렇다면, spontaneous는 관심변수가 아니였는데, 결과변수에 영향을 주고 있으니, 혹시 관심변수(설명변수)에도 영향을 주고 있는
# --> induced와도 관련있다면, confounder일 것이다. induced와의 관계가 궁금해진다(설명, 결과변수 모두 영향시 confounder)
tobj2 <- CreateTableOne(vars = c("induced"),
strata = "spontaneous",
data=dat.i2
)
print(tobj2, test=F) # 이산형 vs 이산형의 비교는 한눈에 잘 안보인다.
tobj3 <- CreateTableOne(vars = c("induce.ever"),
strata = "spontaneous.ever",
data=dat.i2
)
print(tobj3, test=F) # factor(범주형) vs 범주형의 비교는 한눈에 잘 보인다.
# 그러나, row에 범주형 0,1 로 2개가 있을 경우 1개만 보여주는 경향 -> 다보여주게 옵션 줘야한다.
print(tobj3, test=F, showAllLevels=T) # showAllLevels=T
# 해석: strata 칼럼의 범주 종류별로 -> row별로 비율의 차이가 있나 본다.
# -> starat spontaneous.ever 1 : 한번이라도 자염인신중절 했던 그룹 -> (안했던 그룹(52%)에 비해) 인공임신중절(30%)의 비율이 훨씬 낮다))
# --> confounder는 아닐까 의심하고 연구를 시작한다.
# --> 자연임신중절과 인공임신중절은 완전히 독립은 아닐 것이다! 라고 생각해준다.
# - Conditional logistic regression을 쓰려면 clogit()함수를 써야하는데, base아래 없고, survival패키지를 설치해야한다
# - 또한, 회귀분석 결과물을 정리해서 보여주는 broom패키지도 사용한다
library(survival)
library(broom)
# clogit(결과변수~독립변수 + strata(매칭id칼럼) , data=)
cobj <- clogit(case~induce.ever + strata(stratum),
data = dat.i2)
summary(cobj)
# 해석: induce.ever1일 때의, coef(코에피션트)를 보고
# - exp(coef)를 OR로 생각한다.
# - lower .95 upper .95가 OR의 신뢰구간인지 확신할 수 없기 때문에, broom패키지의 tidy( ,exponentiate=T, conf.int=T)함수에 넣어주고 다시 보자.
tidy(cobj, exponentiate = T, conf.int = T)
# estimate : OR
# p.value
# conf.low conf.high
# 해석: induce.ever1(인공임신중절)이, case(1)질병과 아무 관계가 없는 것 같이 보인다
# -> p-value 0.76423 로 독립이라서, 독립변수 - 결과변수 상관관계 X?
# --> 하지만, 독립변수와 비슷해보이는 [자연임신중절]을 confounder로 의심할 수 있었다.
# -> 컨디션 로지스틱회귀에 공변량으로 추가한다.
cobj2 <- clogit(case~induce.ever + spontaneous.ever + strata(stratum),
data = dat.i2)
summary(cobj2)
# 해석1)
# + spontaneous.ever만 추가했을 뿐인데,
# 1) summary()에서 induce.ever1 까지 p-value가 유의해졌다 -> H1:독립X 상관관계 있는 것으로 나온다.
# coef exp(coef) se(coef) z Pr(>|z|)
# induce.ever1 1.0926 2.9821 0.4058 2.693 0.00709 **
# spontaneous.ever1 2.1344 8.4522 0.4050 5.270 1.36e-07 ***
tidy(cobj2, exponentiate = T, conf.int = T)
# induce.ever1의 OR이 3에 가까워졌다(1이면 차이X), p-value도 낮아서 유의하다
# -> [자연임신중절]을 포함시키고 & 고정해놓으면
# --> 관련없어 보이던 [인공임신중절]이 [secondary infertility]와 통계적으로 유의한 관계가 있어진다.
- 코호트 스터디 가기 전 단계
- 많은 증거가 없는 상황에서 시행되므로 high impact 저널에 출판되긴 힘들다.


- 연구질문 :
Sigmoidoscopy(S형 결장내시경)을 이용한 screening이대장암으로 인한 사망을 줄이는가?-
만약, 위험요소 -> 질병의
코호트 or RCT로 분석하면, 대장암이 발병할때까지 기다려야하므로 사실상 무리- 질병 -> 위험요소 노출여부 순으로 분석하는 후향적, Case-Control Study
-
만약, 위험요소 -> 질병의
- 연구대상: Kaiser라는 의료보험 든 사람
-
이렇게 결정한 이유: 위험요소(S형 결장내시경)에 대한 접근이 같아지기 때문
- 소득 수준 등의 차이가 없어진다.
- case : 1971~1987년 사이에 대장암을 진단 받고 1988년 이전에 암으로 사망한 환자
- control : 매칭하여 각 case마다 성별이 같고 /나이와 보험 가입일이 비슷한 (<1년) / 생존자/ 1~4명까지
-
이렇게 결정한 이유: 위험요소(S형 결장내시경)에 대한 접근이 같아지기 때문
- 통계분석 방법
- case, contorl정했으면, 위험요소 노출여부를 언제까지 볼 것인가?
- 생존자료분석이 아니라면, 같은 기간을 보면 된다.(관측기간을 공평하게)
- 매칭 case-control study라서 -> conditional logistic regression을 이용해 OR 등을 구했다.
- adjusted analyses: confounder를 포함한 다변량 모델을 돌렸다
- adjustment는 자기들이 생각한 confounding factors들에 의해 이루어졌다. 그것들을 나열해놓음

- case, contorl정했으면, 위험요소 노출여부를 언제까지 볼 것인가?
- case와 control별, 얼마나
Sigmoidoscopy(위험요소)에 노출되었는지를 디스크립트하게 나타낸 것- 한번도 안받은 사람 / 한번 받은 사람 / 2번이상 받은 사람
- 아무래도 case가 (control에 비해) 위험요소?노출 0회(내시경을 한번도 안받은 사람)의 비율 더 많다
- 다른 검사도, case에 비해 control이 위험요소노출 0회 = 다른 검사를 안받은 비율이 더 높다.
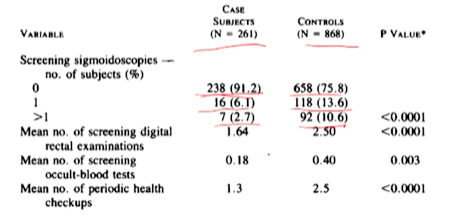
- 다른 검사도, case에 비해 control이 위험요소노출 0회 = 다른 검사를 안받은 비율이 더 높다.
- 핵심 테이블
- 질병, 대장암의 OR를 계산 한 것이 아니라
- S형 내시경을 받을 Odds를 계산 -> case와 control간 ratio(비율)이 달랐는지? OR를 계산했다.
- exposed와 disease를 swtih한 OR^*에 해당하는 값이다.
-
원래 생각하던 OR과 값이 같으니 해석도 -> 대장암의 OR로 해석하면 된다.
- 대장암뿐만 아니라, 내시경확인불가 cancer를 포함하여 나눠서 계산했다
- OR이 1보다 작다 ->
대장암 환자일수록 -> 내시경을 전에 받았을 OR가 더 낫다 -
반대로
내시경을 전에 더 많이 받았을 수록 -> 대장암일 OR가 더 낫다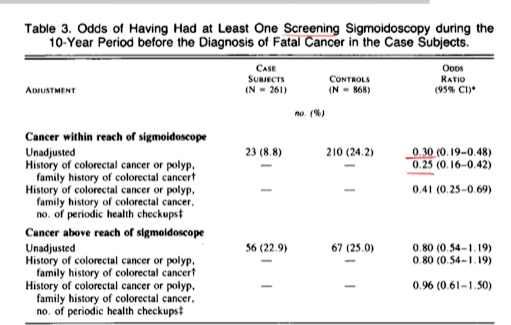
- Case-control study는 질병을 가지고 있는 환자군(cases)을 먼저 선정하고, 그 질병이 없는 대조군(controls)을 선정하여, 두 그룹 간 과거의 위험요소 노출여부나 특성을 비교하는 연구이다
- Case-control study에서는 RR은 직접 추정할 수 없으나, OR은 추정 가능하다
- 매칭하지 않은 case-control study의 OR 추정방법과 confounding, interaction을 다루는 방법은 cohort study와 거의 같다
- Matched case-control study의 OR 추정은 M-H 방법이나 conditional logistic regression을 이용한다
- 관찰연구의 일종이다.
- 실험연구 아니면 다 관찰연구
- 여러개의 질병을 동시에 연구할 수 없다.
- 질병 먼저 -> 뒤쪽에 시간들여서 찾는 위험요소노출이 여러개일 수 있다. 질병은 1개
- 희귀병이나, 위험요소 노출부터 질병 발병까지의 기간이 너무 긴 질병의 연구에 적합하다.
- 오래기다려야하는 병들을 미리 찍어서 과거를 보러 간다
- prevalence에 대한 추론이 가능하다.
- 연구자가 case 정하고 -> control을 그에 맞춰 1~4명까지 정한다 -> 모집단과는 전혀다르게 prevalence가 뻥튀기 된다.
- 정답: 4
- 해설: Case-control study에서는 case와 control의 비율이 연구자에 의해 인위적으로 조정되므로, prevalence에 대한 추론을 할 수 없다.
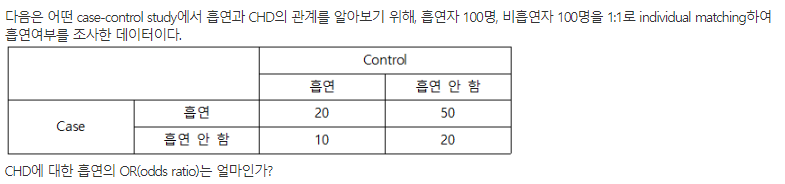
- 주어진 데이터 자료구조를 보고
-
cohort studyorunmatched case-control study문제인지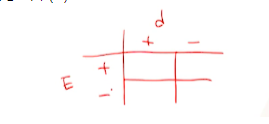
-
matched case-conrol study문제 인지 판단한다
-
- matched case-conrol study문제의 OR은 n12/n21로 구한다
- n12 = case(앞) exposed(1) / control(뒤) unexposed(2)
- n21 = case(앞) unexposed(2) / control(뒤) exposed(1)
- 50/10 = 5.0